Deep-Learning-basierte Klassifikation von histologischen Subtypen von Lungentumoren
Deep-Learning-basierte Klassifikation von histologischen Subtypen von Lungentumoren
Deep-Learning-Methoden (hier Convolutional Neural Networks) können anhand von Bildern histologischer Schnitte verschiedene Arten Lungentumoren ähnlich gut wie ein Pathologe unterscheiden.
Die Einteilung von Lungentumoren in verschiedene Subtypen ist ein wichtiges Kriterium zur Wahl der geeigneten Behandlung von Lungenkrebspatienten. Im klinischen Alltag wird dies im Moment durch trainierte Pathologen durchgeführt, indem histologische Schnitte mit H&E-Färbung in einem optischen Mikroskop visuell inspiziert werden.
In dieser Studie diskriminieren wir zwischen den beiden häufigsten Typen von Lungenkrebs: Adenokarzinom und Plattenepithelkarzinom. Wir haben von 207 Patienten mit bekannter Diagnose Bilddaten, wobei von jedem Patientensample 50 histologische Bilder vorliegen.
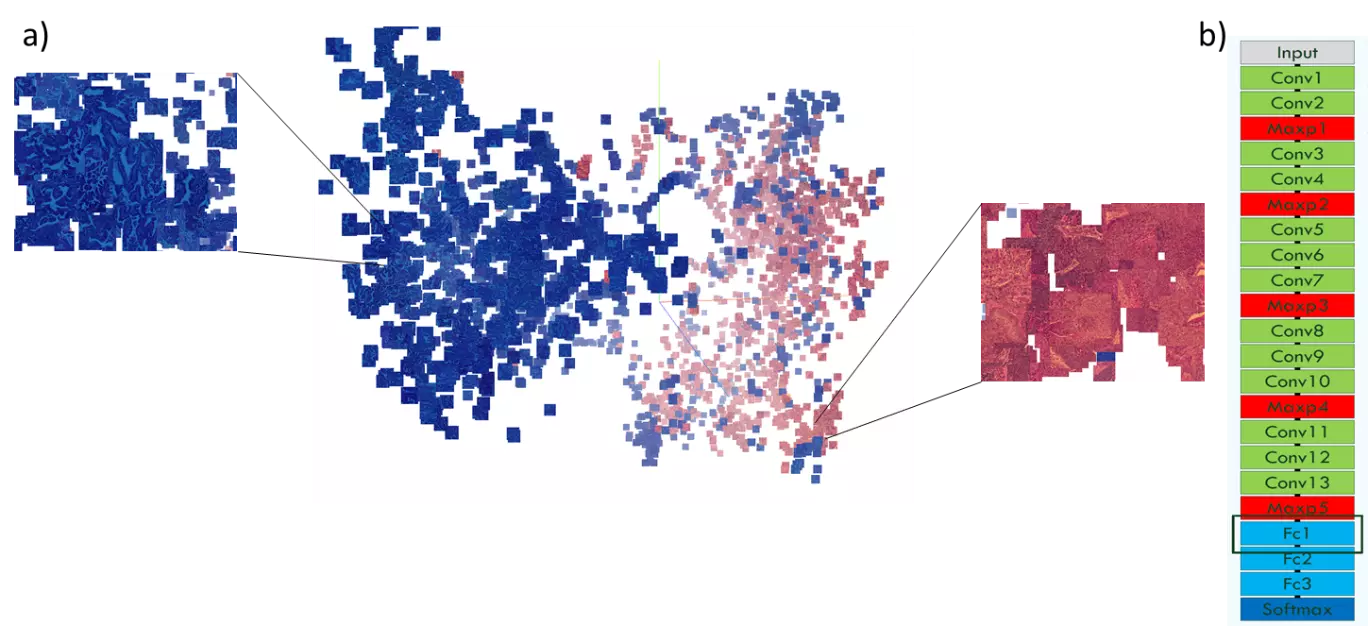
b) Die VGG-Architektur des vortrainierten CNN, das benutzt wurde, um aus jedem Bild im markierten Layer die 4096 Merkmale zu extrahieren.
In einem ersten Schritt haben wir ein vortrainiertes CNN mit VGG-Architektur benutzt, um aus den Bildern Merkmale zu extrahieren. Dieses vortrainierte VGG wurde im Rahmen des ImageNet Wettbewerbs auf ca 1 Million Bildern trainiert, um zwischen 1000 verschiedene Klassen (z.B Containerschiff, Leopard, Pilz etc.) zu diskriminieren. Wir benutzen die histologischen Schnitte als Eingabebilder für dieses vortrainierte VGG-Netz (Bild rechts) und extrahieren die Aktivierungen in einem der letzten Layer vor der Klassifikation, die dann als neue Merkmalsdarstellung der Bilder dienen. Diese so erzeugten Merkmale dienen als Grundlage für eine Anordnung der Bilder in einer 3D t-SNE Karte (Bild links). In dieser t-SNE Darstellung ist bereits eine deutliche Trennung der beiden Tumortypen zu erkennen, was darauf schliessen lässt, dass die Bildmerkmale geeignet sind, um Strukturen in den Bildern zu beschreiben, durch die sich die beiden Tumortypen unterscheiden. Dabei ist zu beachten, dass in keinem Schritt (ausser in der farblichen Markierung in t-SNE Karte), die Klassenzugehörigkeit der histologischen Schnitte benutzt wurden!
Wir können bereits in einer solchen t-SNE Darstellung eine Nächste-Nachbar Klassifikation verwenden, um Bilder von einem unbekannten Tumortyp, einem der beiden Klassen zuzuordnen. Wir können diese neuen Merkmale aber auch zum Trainieren eines anderen Klassifikators verwenden, wie beispielsweise einem Random Forest oder einem Neuronalen Netz. Wir können aber auch ein CNN «from scratch» mit den vorhandenen histologischen Bildern mit bekannter Diagnose trainieren, wobei wir einige Tricks wie beispielsweise Daten-Augmentierung anwenden müssen, um mit der begrenzten Anzahl von Bildern auszukommen. Die beste Klassifikationsperformance erzielen wir jedoch, wenn wir vom vortrainierten VGG starten, und nur den letzten Teil des Netzes nachtrainieren («finetuning») und so einerseits die Generierung der Merkmale ans neue Problem anzupassen und andererseits die Kombination der angepassten Merkmale zur Vorhersage der Wahrscheinlichkeit für die beiden Tumortypen zu lernen. Mit einem solchen Verfahren zeigt dieses automatisierte Klassifikationsverfahren eine ähnliche Performance wie trainierte Pathologen. Das System ist inzwischen im Unispital Zürich zur Unterstützung der Pathologen im Einsatz.