Forschungsschwerpunkt Computational Health
Der Forschungsschwerpunkt Computational Health befasst sich mit Fragestellungen aus Medizin und Biologie unter Verwendung datengetriebener und mechanistischer Modellierung. Wichtige Instrumente sind maschinelles Lernen für die Bild- und Signalanalyse, graphische Netzwerke, Parameterschätzung für Differenzialgleichungssysteme und physiologische Simulation.
Über uns
Der Schwerpunkt Computational Health befasst sich mit fundamentalen Fragestellungen aus Biologie und Medizin unter Verwendung computergestützter, datengetriebener Methoden. Wichtige Instrumente sind maschinelles Lernen für die Bild- und Signalanalyse, Parameterschätzung für Differenzialgleichungssysteme und Multiphysik-Simulation. Effektive Validierungsstrategien werden eingesetzt, um mit dem Unbekannten umzugehen.
Unsere Forschungsgruppen
Biomedical Simulation
Die Forschungsgruppe ist spezialisiert auf die Modellierung biologischer und medizinischer Systeme. Es werden neue Ansätze entwickelt zur Simulation physiologischer Prozesse und um krankhafte Veränderung vorherzusagen. Insbesondere fliesst vertieftes Wissen zu biologischen/physiologischen Prozessen in Multi-Physik Simulationen ein.
Die Gruppe entwickelt Algorithmen zur Parameter- und Unsicherheitsschätzung physikalisch motivierter stochastischer Modelle. Insbesondere werden Maschinelle Lernverfahren mit Bayes’scher Modellierung verknüpft zur Reduktion der Dimensionalität. Diese Verfahren finden breite Anwendung in der Medizin und in den Life Sciences.
Leiter: Prof. Dr. Sven Hirsch | Erfahren Sie mehr über die Forschungsgruppe Biomedical Simulation
Medical Image Analysis
Die Forschungsgruppe wendet maschinelle Lernverfahren an zur Interpretation medizinischer Bilddaten. Derart werden Merkmale extrahiert zur Charakterisierung von Krankheitsbildern und zur Verwendung als diagnostische Marker. Von besonderem Interesse sind die radiomische und morphologische Vermessung diagnostischer Bilddaten. Hierbei verfolgt die Gruppe das Ziel, mittels erklärbarer maschineller Lernverfahren reproduzierbare, bildbasierte Biomarker zu etablieren und deren klinischen Nutzen sicherzustellen.
Medical Data Modelling
Die Forschungsgruppe wendet Verfahren der Statistik und des maschinellen Lernens an zur Modellierung von Wirkzusammenhängen in medizinischen Daten, um insbesondere pathophysiologische Prozesse zu analysieren. Patientendaten werden zusammen mit Daten aus Bildgebungsverfahren, insbesondere der Magnetresonanztomographie, analysiert.
Biosignal Analysis & Digital Health
Die Forschungsgruppe untersucht Daten von Wearables und Biosensoren mit Methoden der Zeitreihenanalyse und verbindet diese mit biologisch-physikalischen Modellen zur robusten Charakterisierung von physiologischen Zuständen. Diese Datenquellen dienen zur Umsetzung von Patient Reported Outcomes in die klinische Praxis und zur Weiterentwicklung der patientenzentrierten Medizin.
Leiter: Dr. Samuel Wehrli | Erfahren Sie mehr über die Forschungsgruppe Biosignal Analysis & Digital Health
Lehre
Das Lehrangebot des Forschungsschwerpunktes Computational Health umfasst Teilgebiete der mathematischen und physikalischen Modellierung. Auf Stufe Bachelor ist sie verantwortlich für die Vertiefung Digital Health. In weiteren Bachelor Lehrgängen am Departement unterrichtet sie Physik und Mathematik. Im MSc Applied Computational Life Sciences und im PhD-Programm Data Science bieten sie vertiefte Kurse zur Modellierung komplexer Systeme an.
Team Computational Health
Projekte
- Seite 01
- Seite 02
- Seite 03
- Seite 04
- Nächste Seite
-
Digital Health Zurich – Ein Praxislabor für patientenzentrierte klinische Innovation
Digital Health Zurich erforscht digitale Gesundheitslösungen im Spitalkontext und setzt diese effizient und praxisrelevant um. Kernthemen sind PROMs (Patient Reported Outcome Measure), Fernüberwachung, integrierte Pflege und damit verbundene Technologien sowie die Befähigung von Patient:innen und Personal. Unsere ...
-
Modellierung multizentrischer und dynamischer Schlaganfall Gesundheitsdaten
Ziel des Projektes ist die Weiterentwicklung von probabilistischer und v.a. dynamischer Modellierung von Krankheiten in Form von Bayes’schen Netzwerken im Bereich der Digitalen Gesundheit als zentralen strategischen Pfeiler der neu formierten Fachgruppe «Medical Image Analysis and Data Modeling» des Schwerpunktes ...
-
Feature Learning for Bayesian Inference
The goal of this project is to use interpretable Machine Learning (ML) to find low-dimensional features in high-dimensional noisy data generated by (i) stochastic models or (ii) real systems. In both cases, the problem is to disentangle the effect of high-dimensional disturbances, such as noise or unobserved inputs, ...
-
OSR4H – Open Set Recognition for Hematology
Development of a Proof of Concept for visual Open Set Recognition (OSR) algorithms applied to a Hematology task, the classification of white blood cells.
-
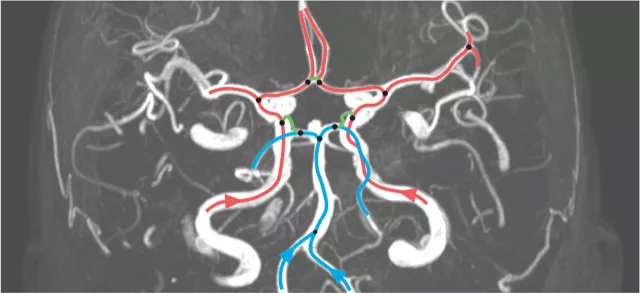
Deep Brain Vessel Profiler
Es wird vermutet, dass die Architektur der versorgenden Hirngefässe das Auftreten und den Schweregrad häufiger zerebrovaskulärer Erkrankungen wie ischämische Schlaganfälle oder intrakranielle Aneurysmen beeinflusst. In diesem Projekt untersuchen wir Methoden zur effizienten Quantifizierung der Variabilität in ...
Publikationen
-
Juchler, Norman; Schilling, Sabine; Bijlenga, Philippe; Kurtcuoglu, Vartan; Hirsch, Sven,
2022.
Frontiers in Neurology.
13(809391).
Verfügbar unter: https://doi.org/10.3389/fneur.2022.809391
-
Suter, Susanne; Spinner, Georg; Hoelz, Bianca; Rey, Sofia; Thanabalasingam, Sujeanthraa; Eckstein, Jens; Hirsch, Sven,
2022.
Visualization and analysis of wearable health data from COVID-19 patients.
arXiv.
Verfügbar unter: https://doi.org/10.21256/zhaw-24219
-
Delucchi, Matteo; Spinner, Georg Ralph; Scutari, Marco; Bijlenga, Philippe; Morel, Sandrine; Friedrich, Christoph M.; Hirsch, Sven,
2022.
Bayesian networks to disentangle the interplay of intracranial aneurysm rupture risk factors [Paper].
In:
Nithiarasu, Perumal; Vergara, Christian, Hrsg.,
CMBE 2022 : 7th International Conference on Computational & Mathematical Biomedical Engineering.
7th International Conference on Computational and Mathematical Biomedical Engineering (CMBE22), Milan, Italy, 27-29 June 2022.
Computational and Mathematical Biomedical Engineering.
S. 22-25.
-
Ulzega, Simone; Albert, Carlo,
2022.
In:
EGU General Assembly 2022, Vienna, Austria, 23-27 May 2022.
Verfügbar unter: https://doi.org/10.5194/egusphere-egu22-8729
-
Dupuy, Nicolas; Juchler, Norman; Morel, Sandrine; Kwak, Brenda R.; Hirsch, Sven; Bijlenga, Philippe,
2022.
Exploring intracranial aneurysm instability markers to improve disease modeling [Paper].
In:
Nithiarasu, Perumal; Vergara, Christian, Hrsg.,
CMBE 2022 : 7th International Conference on Computational & Mathematical Biomedical Engineering.
7th International Conference on Computational and Mathematical Biomedical Engineering (CMBE22), Milan, Italy, 27-29 June 2022.
Computational and Mathematical Biomedical Engineering.
S. 14-17.
Verfügbar unter: https://doi.org/10.21256/zhaw-25387










