Forschungsschwerpunkt Computational Health
Der Forschungsschwerpunkt Computational Health befasst sich mit Fragestellungen aus Medizin und Biologie unter Verwendung datengetriebener und mechanistischer Modellierung. Wichtige Instrumente sind maschinelles Lernen für die Bild- und Signalanalyse, graphische Netzwerke, Parameterschätzung für Differenzialgleichungssysteme und physiologische Simulation.
Über uns
Der Schwerpunkt Computational Health befasst sich mit fundamentalen Fragestellungen aus Biologie und Medizin unter Verwendung computergestützter, datengetriebener Methoden. Wichtige Instrumente sind maschinelles Lernen für die Bild- und Signalanalyse, Parameterschätzung für Differenzialgleichungssysteme und Multiphysik-Simulation. Effektive Validierungsstrategien werden eingesetzt, um mit dem Unbekannten umzugehen.
Unsere Forschungsgruppen
Biomedical Simulation
Die Forschungsgruppe ist spezialisiert auf die Modellierung biologischer und medizinischer Systeme. Es werden neue Ansätze entwickelt zur Simulation physiologischer Prozesse und um krankhafte Veränderung vorherzusagen. Insbesondere fliesst vertieftes Wissen zu biologischen/physiologischen Prozessen in Multi-Physik Simulationen ein.
Die Gruppe entwickelt Algorithmen zur Parameter- und Unsicherheitsschätzung physikalisch motivierter stochastischer Modelle. Insbesondere werden Maschinelle Lernverfahren mit Bayes’scher Modellierung verknüpft zur Reduktion der Dimensionalität. Diese Verfahren finden breite Anwendung in der Medizin und in den Life Sciences.
Leiter: Prof. Dr. Sven Hirsch | Erfahren Sie mehr über die Forschungsgruppe Biomedical Simulation
Medical Image Analysis
Die Forschungsgruppe wendet maschinelle Lernverfahren an zur Interpretation medizinischer Bilddaten. Derart werden Merkmale extrahiert zur Charakterisierung von Krankheitsbildern und zur Verwendung als diagnostische Marker. Von besonderem Interesse sind die radiomische und morphologische Vermessung diagnostischer Bilddaten. Hierbei verfolgt die Gruppe das Ziel, mittels erklärbarer maschineller Lernverfahren reproduzierbare, bildbasierte Biomarker zu etablieren und deren klinischen Nutzen sicherzustellen.
Medical Data Modelling
Die Forschungsgruppe wendet Verfahren der Statistik und des maschinellen Lernens an zur Modellierung von Wirkzusammenhängen in medizinischen Daten, um insbesondere pathophysiologische Prozesse zu analysieren. Patientendaten werden zusammen mit Daten aus Bildgebungsverfahren, insbesondere der Magnetresonanztomographie, analysiert.
Biosignal Analysis & Digital Health
Die Forschungsgruppe untersucht Daten von Wearables und Biosensoren mit Methoden der Zeitreihenanalyse und verbindet diese mit biologisch-physikalischen Modellen zur robusten Charakterisierung von physiologischen Zuständen. Diese Datenquellen dienen zur Umsetzung von Patient Reported Outcomes in die klinische Praxis und zur Weiterentwicklung der patientenzentrierten Medizin.
Leiter: Dr. Samuel Wehrli | Erfahren Sie mehr über die Forschungsgruppe Biosignal Analysis & Digital Health
Lehre
Das Lehrangebot des Forschungsschwerpunktes Computational Health umfasst Teilgebiete der mathematischen und physikalischen Modellierung. Auf Stufe Bachelor ist sie verantwortlich für die Vertiefung Digital Health. In weiteren Bachelor Lehrgängen am Departement unterrichtet sie Physik und Mathematik. Im MSc Applied Computational Life Sciences und im PhD-Programm Data Science bieten sie vertiefte Kurse zur Modellierung komplexer Systeme an.
Team Computational Health
Projekte
- Vorherige Seite
- Seite 01
- Seite 02
- Seite 03
- Seite 04
- Nächste Seite
-
Data mining in neurological medicine
Restless legs syndrome (RLS, Willis-Ekbom disease) is a neurological movement disorder characterised by motor and sensory symptoms, such as the uncontrollable need to move the legs (and sometimes also the arms). Such need is associated with an unpleasant and disturbing sensation in the lower limbs that typically ...
-
Digitale Simulation zur individualisierten Fertigung von 3D Nanofaserfilter und Integration in Vollschutzanzug für Pandemiefälle
We intend the tailored fabrication of 3D nanofiber aerogel (NFA) particle filters and their integration in a full protective suite with blower device for the health market. Digital simulation of the particle nanofiber interaction is used to predict the best microscopic filter geometry.
-
Platform Health
Researchers from the School of Life Sciences and Facility Management (LSFM) initiated a platform to promote interdisciplinary research in the field of health. The School of LSFM supports this initiative to increase visibility of all health-research related activities in teaching, R&D, continued education, and ...
-
Radiosands
Radiosands ist eine begehbare Installation, in der mehrere gleichzeitig stattfindende Radiosendungen analysiert und neu zusammengesetzt werden. Die Installation nutzt die Geschwindigkeit und algorithmische Potenz digitaler Technik, um eine neue Erfahrung zu schaffen: Eine Echtzeit-Collage von klanglichen Fragmenten, ...
-
BISTOM - Bayesian Inference with Stochastic Models
In essentially all applied sciences, data-driven modeling heavily relies on a sound calibration of model parameters to measured data for making probabilistic predictions. Bayesian statistics is a consistent framework for parameter inference where knowledge about model parameters is expressed through probability ...
Publikationen
-
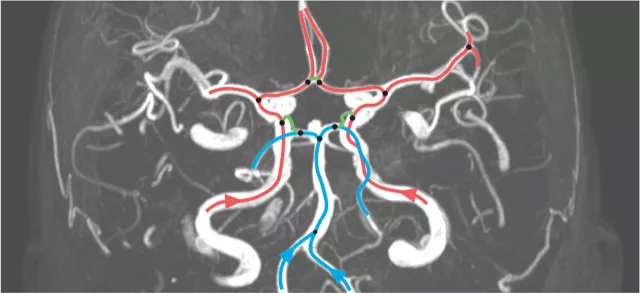
Musio, Fabio; Juchler, Norman; Yang, Kaiyuan; Menze, Björn; Hirsch, Sven,
2023.
Circle of Willis configurations in stroke patients.
In:
19th Interdisciplinary Cerebrovascular Symposium, Geneva, Switzerland, 17-19 August 2023.
-
Reissenberger, Pamela; Serfözö, Peter; Piper, Diana; Juchler, Norman; Glanzmann, Sara; Gram, Jasmin; Hensler, Karina; Tonidandel, Hannah; Börlin, Elena; D’Souza, Marcus; Badertscher, Patrick; Eckstein, Jens,
2023.
European Heart Journal - Digital Health.
4(5), S. 402-410.
Verfügbar unter: https://doi.org/10.1093/ehjdh/ztad039
-
Bächinger, David; Filidoro, Noemi; Naville, Marc; Juchler, Norman; Kurtcuoglu, Vartan; Nadol, Joseph B.; Schuknecht, Bernhard; Kleinjung, Tobias; Veraguth, Dorothe; Eckhard, Andreas H.,
2023.
Scientific Reports.
13(1), S. 10303.
Verfügbar unter: https://doi.org/10.1038/s41598-023-36479-5
-
Ulzega, Simone; Albert, Carlo; Beer, Jürg,
2023.
In:
5th Swiss SCOSTEP Workshop, Windisch, Switzerland, 15-16 May 2023.
Verfügbar unter: http://scostep2023.cs.technik.fhnw.ch/pres/ulzega_scostep_2023.pdf
-
Bacci, Marco; Sukys, Jonas; Reichert, Peter; Ulzega, Simone; Albert, Carlo,
2023.
A comparison of numerical approaches for statistical inference with stochastic models.
Stochastic Environmental Research and Risk Assessment.
Verfügbar unter: https://doi.org/10.1007/s00477-023-02434-z










